推定で、5万とも10万ともいわれる全ての遺伝子と、それらの間に介在する領域の全ての塩基を解き明かすこと−すなわち、ヒトゲノム(47)の地図の作製−は、15年もの歳月を掛けた国際協力事業ヒトゲノムプロジェクト(48)として知られています。(訳者注;2001年に既に全染色体の読み込みが終了し、現在ではヒトの遺伝子の数は約32,000であるとされています)。科学者は、人間の遺伝子の完全な詳細地図を手に入れることで、医療や生物医学研究が大きく変わっていくことを期待しています。
ヒトゲノムプロジェクトは、ゲノム地図(49)の作製の中でも特に遺伝子の相対的位置関係を示す遺伝子連鎖地図(50)(genetic linkage maps)と実際の染色体領域に直接マップする物理的地図(51)(physical maps)の二つに焦点を当てています。ゲノム地図とはマーカー遺伝子(52)あるいは、その他の指標となる遺伝子が染色体中のどの位置にあるかを示すものです。
遺伝連鎖地図はある遺伝子の固有の短い配列がマーカーのどの位地にあるかを示すもので、マーカー遺伝子同士がどのくらいの確率で一緒に遺伝していくのかというデータに基づき作成されたものです。この連鎖地図は遺伝子組み替え(53)または交叉(54)と呼ばれる現象を利用したものです。精子と卵子が作られていく過程で、対になった母親または父親由来の染色体の一部が入れ替わることがあります。遺伝子とその近くにあるマーカー遺伝子は、遠い位置関係にあるマーカー遺伝子同士が組み替えの際にばらばらになる確率と比べて、かなり高い確率で一緒に行動をとることになります。1994年各国の研究者が合同で5000種以上ものマーカーと400種以上もの遺伝子の包括的な連鎖地図を発表しました。
遺伝子地図を利用するようになって、研究者は遺伝子の位置を染色体上の比較的小さな領域の中で表現できるようになりました。次の段階として、さらに細かい位置を決めるために、現在物理的地図の作成作業に取りかかっています。
物理的地図の構築のためには、まず染色体(場合によっては、全ゲノム)をまずDNAの小片に壊し、複製・クローン(55)化し、同一の遺伝子断片のコピーを何百万個も作ります。次に、得られたクローンを元の染色体上の順番に従って並べ替えます。そして、その場所と得られた遺伝子解析結果と染色体上での順序(これを隣接(56)と呼んでいます)をコンピューターに記録し、クローンそのものは研究室のフリーザーに保管します。遺伝子連鎖地図で、ある遺伝子が特定の場所に位置することがわかれば、研究者はフリーザーからそのクローンを取り出し、目的の遺伝子を同定し、そのクローンを材料として行います。DNAシークエンス(57)とは遺伝子のすべての塩基の配列を決定する方法です。
詳細な物理的地図と高性能のDNAシークエンス技術・機器の進歩により、新しい疾患関連遺伝子が月に数個の割合で物理学的地図に書き込まれ、さらに単離されています。2005年までには、5万から10万にも上るといわれる遺伝子の全ての位置が正確に突き止められ、塩基の正確な配列が確認できるという希望のもと、研究が進められています(訳者注;これまでの研究から、ヒトの遺伝子の数は約32,000であることがわかりました)。
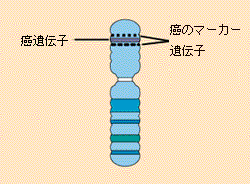
図13:特定の遺伝子を絞り込むために、研究者は遺伝子マーカー―DNAや遺伝子のすでに知られている特徴を表すもの―の同定を行います。遺伝子マーカーは目的遺伝子のすぐ近くに位置し、その遺伝子とともに受け継がれます。
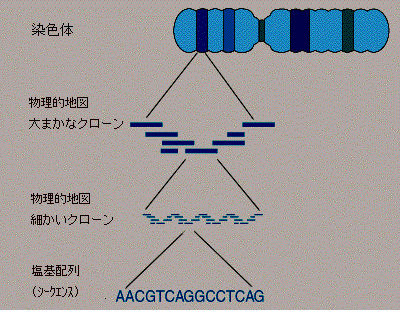
図14:DNA地図はその詳しさによりいくつかの段階があります。具体的には、染色体の特定の色素で染めたときに見える縞模様で判別する大まかな地図、ある目的の遺伝子と相同的な遺伝子を含むDNAを示した地図、そしてDNAの塩基配列を示す地図があります。(これはちょうど、世界地図、日本地図から、住宅地図までの段階があるのと同様です。)
|